学会这3张图,搞定数据质控Soeasy
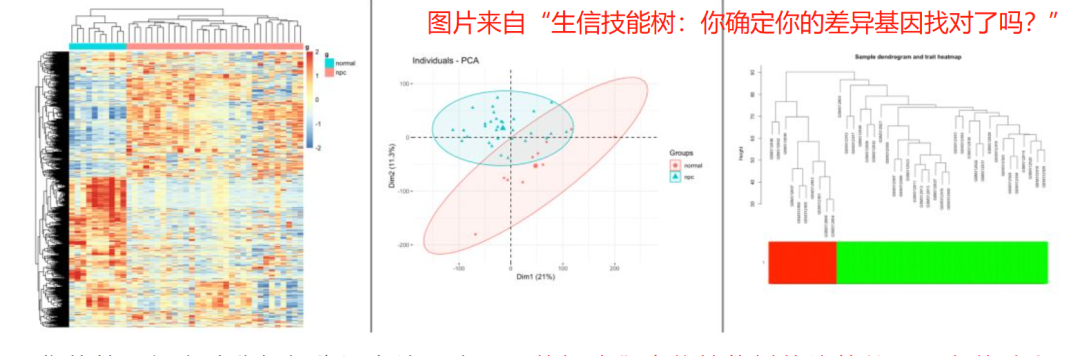
嗨,小伙伴们大家好~!上期我们讲到数据质控的重要性,即在表达差异分析之前需查验数据质量是否合格,如下面3张图所示,热图、PCA图和聚类树的结果均表示实验组和对照组分开的比较好,数据质量可。那么这期我们就接着这个话题,来看看如何绘制这3张图,搞定数据质控问题Soeasy!
▌加载示例数据
加载基因表达谱和样本信息文件,geneexpres是已完成基因注释和标准化处理的基因表达谱,sample_info是样本信息,其中包含1个干预组(AceP)和1个对照组(NC),每组包含3个样本(AceP1~3;NC1~3),每个样本是一个批次(AceP1/NC1;AceP1/NC1;AceP1/NC1),添加batch信息。
library(tidyverse)head(gene_exp_Ace)(sample_Ace)AceP_1AceP_1AcePAceP_3AceP_3AcePNC_2NC_2Control样本添加batch信息sample_Ace-sample_Ace%%mutate(batch=paste0("batch",c(1,2,3,1,2,3)))sample_AceAceP_1AceP_1AcePbatch1AceP_3AceP_3AcePbatch3NC_2NC_2Controlbatch2▌数据质量评价
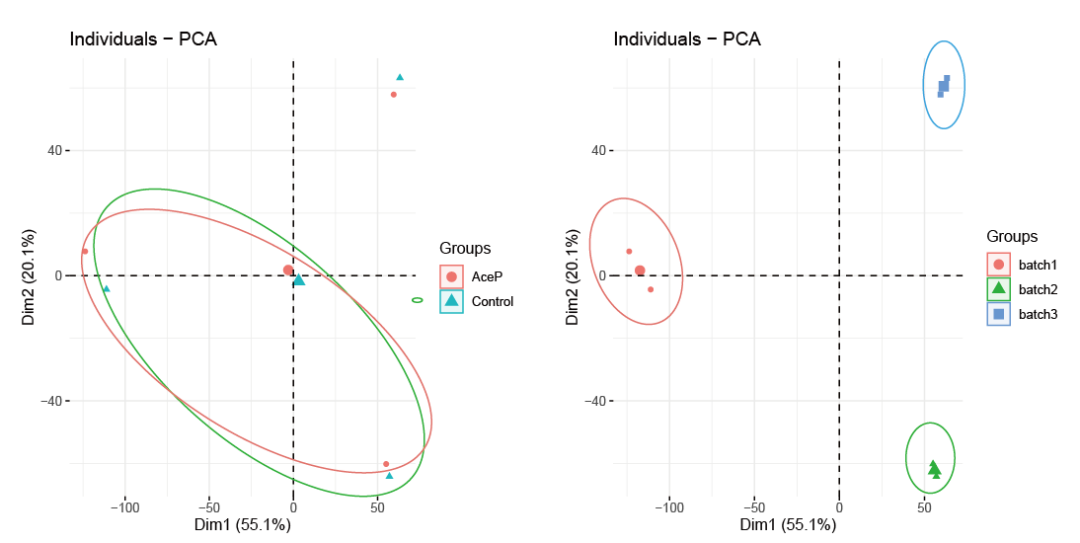
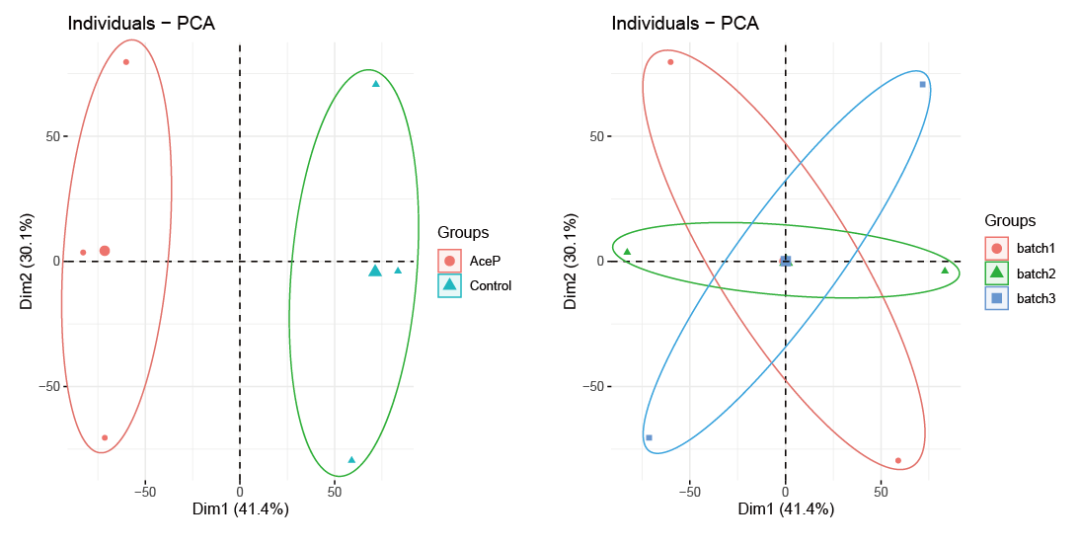
第一张图:PCA图,使用fvizpcaind函数。PCA直观可以看到干预组和对照组完全没有分开,样本是按照3个批次来聚类的,数据存在很明显的批次效应。
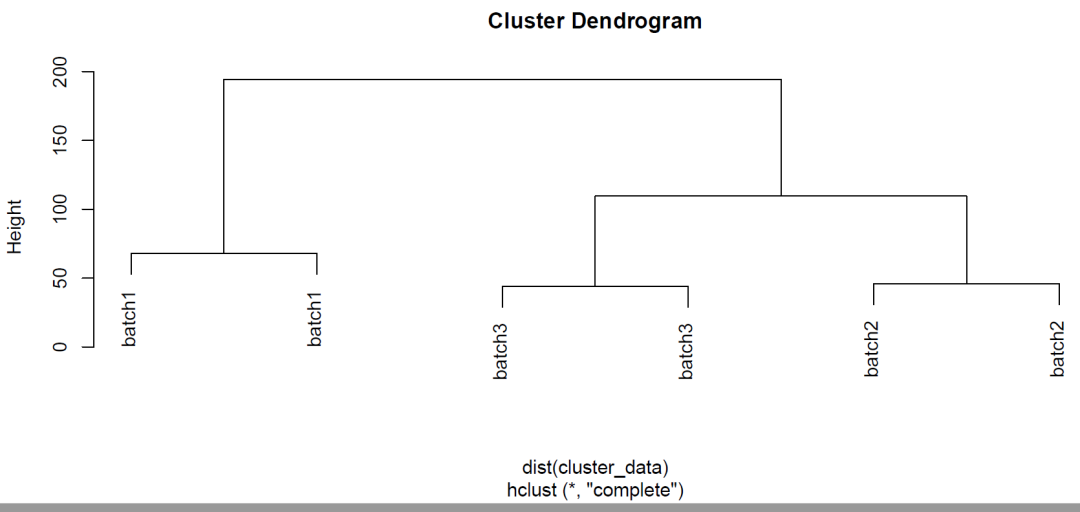
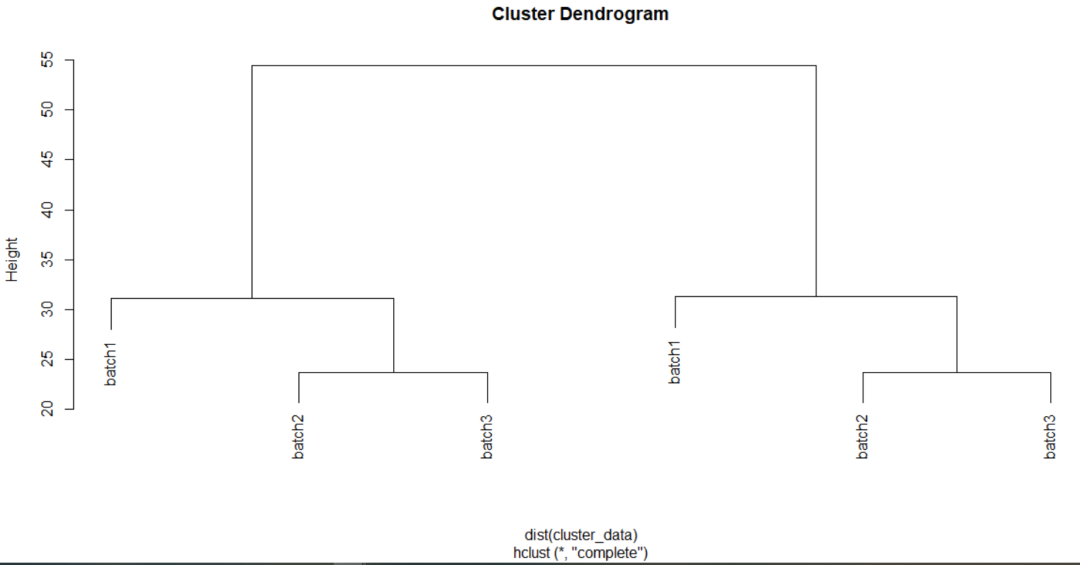
按分组标注fviz_pca_ind(expr_pca,label="none",habillage=pca_data$group_res,按批次标注fviz_pca_ind(expr_pca,label="none",habillage=pca_data$batch,第二张图:聚类树图按batch聚类cluster_data-t(gene_exp_Ace)rownames(cluster_data)=sample_Ace$batchhc=hclust(dist(cluster_data))plot(hc)
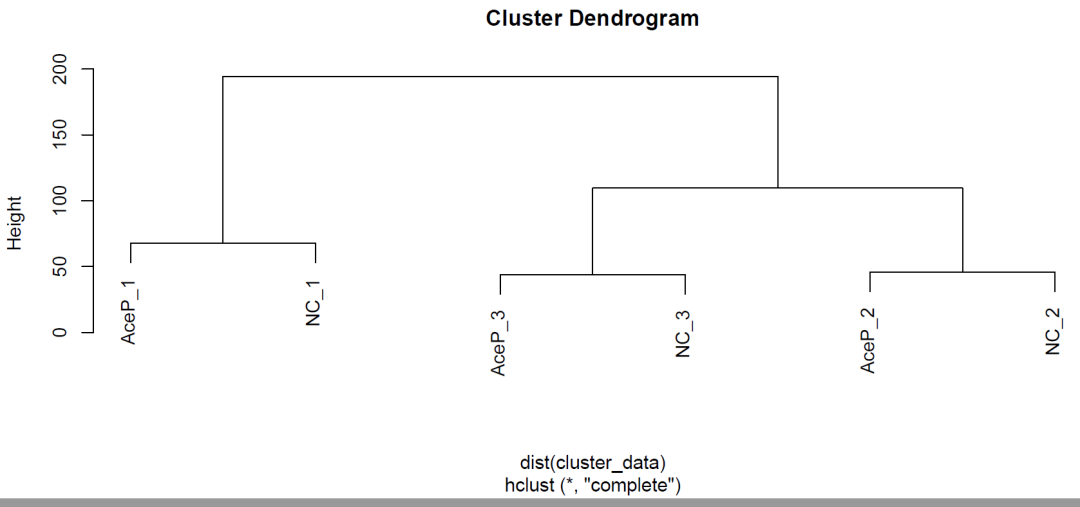
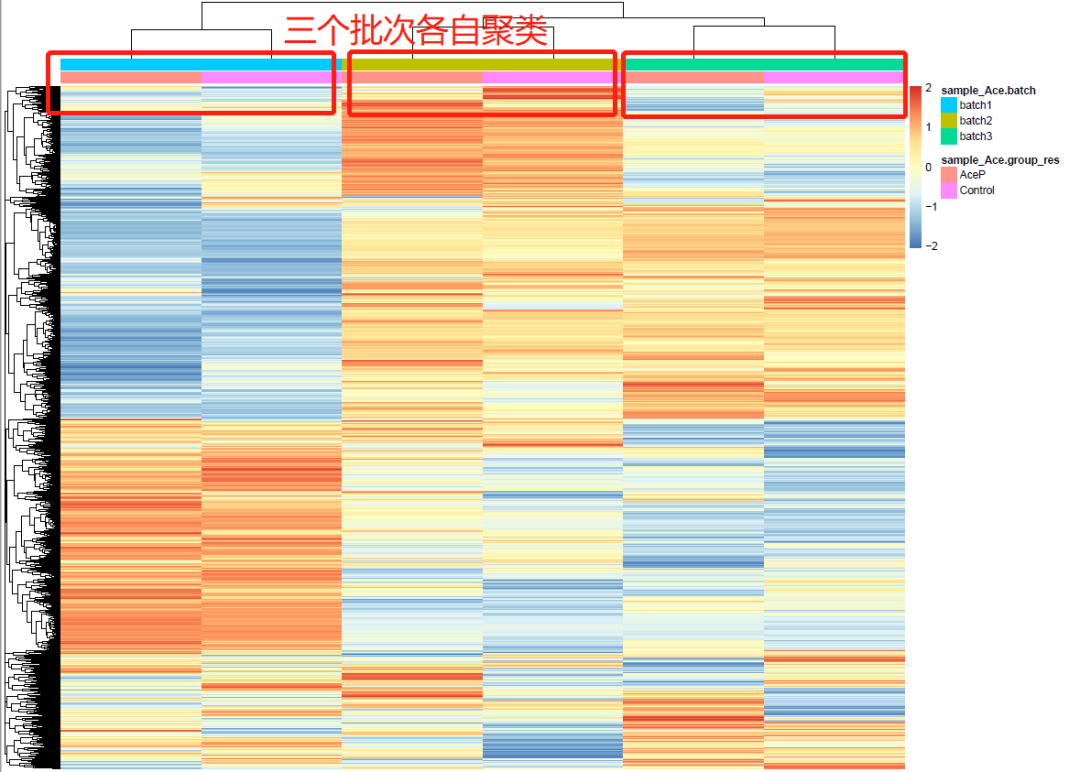
第三张图:聚类热图,pheatmap函数实现。聚类热图也是同样的结果,直观可见干预组和对照组的样本是按照3个批次来聚类的。
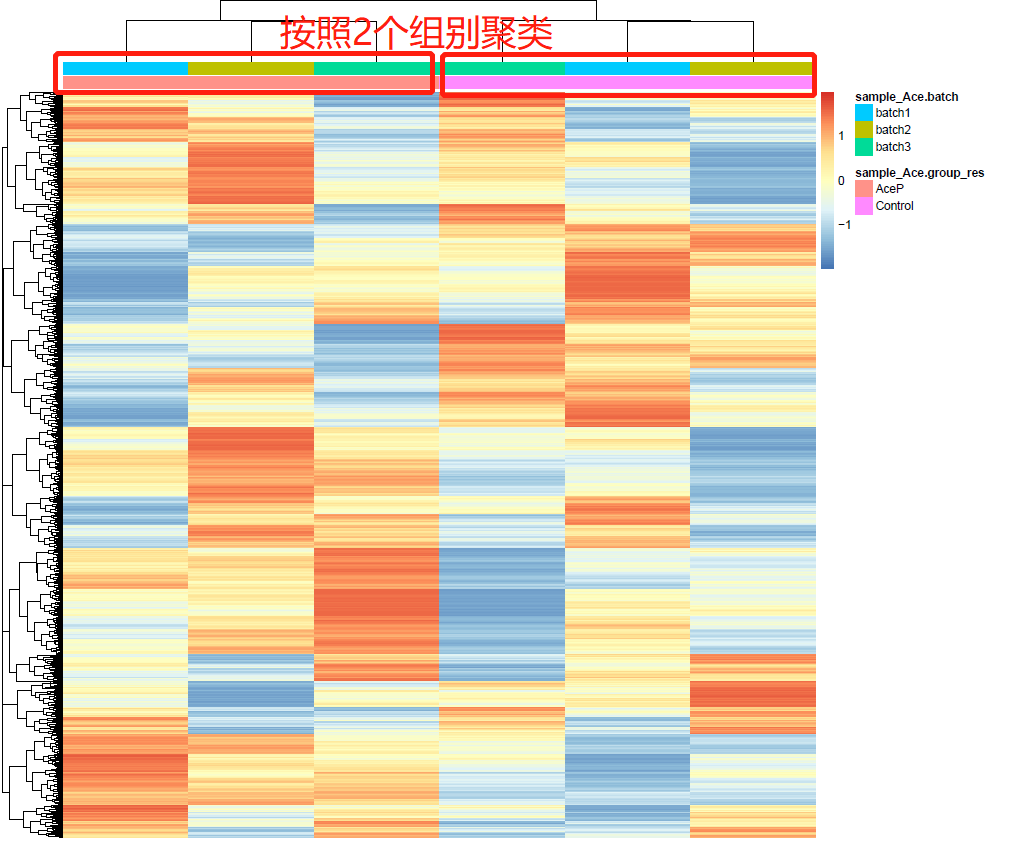
列注释信息annotation_(sample_Ace$group_res,sample_Ace$batch)rownames(annotation_col)-colnames(gene_exp_Ace)去除批次效应ComBatFound3batchesStandardizingDataacrossgenesFindingparametricadjustments第一张图:PCA图fviz_pca_ind函数expr_pca-t(exprAce_batch)pca_data-cbind(expr_pca,sample_Ace)expr_pca-prcomp(pca_data[,1:15061],scale=TRUE)expr_pcalibrary(factoextra)按分组标注addEllipses=TRUE,=0.95)按批次标注addEllipses=TRUE,=0.95)按样本聚类cluster_data-t(exprAce_batch)hc=hclust(dist(cluster_data))plot(hc)第三张图:聚类热图绘制热图library(pheatmap)pheatmap(exprAce_batch,scale="row",show_colnames=F,show_rownames=F,border_color=NA,annotation_col=annotation_col,annotation_names_col=F)

总结
汇总一下去除批次效应前后的3张图,大家细品~!
好啦,以上就是今日推文的全部内容了,小伙伴们我们下期再见~!







